乳腺癌每年新发病例已达230万!这5类乳腺癌小鼠模型值得关注
乳腺癌作为全球女性发病率最高的恶性肿瘤,其防治挑战备受关注。国际癌症研究机构(IARC)数据显示,2022年全球乳腺癌新发病例达230万(占女性癌症新发病例的25%),死亡病例67万(占女性癌症死亡的15.5%)。已超越肺癌成为女性第一大癌症[1]。我国乳腺癌现状更为严峻:发病率增速远超世界平均水平,且患者平均确诊年龄仅46岁[2],中位确诊年龄为45-55岁,年轻化趋势显著[2-3]。今天,亚星yaxing868官网将系统解析乳腺癌的临床分期分型体系,并重点介绍乳腺癌研究中常用的小鼠模型类型及应用价值。
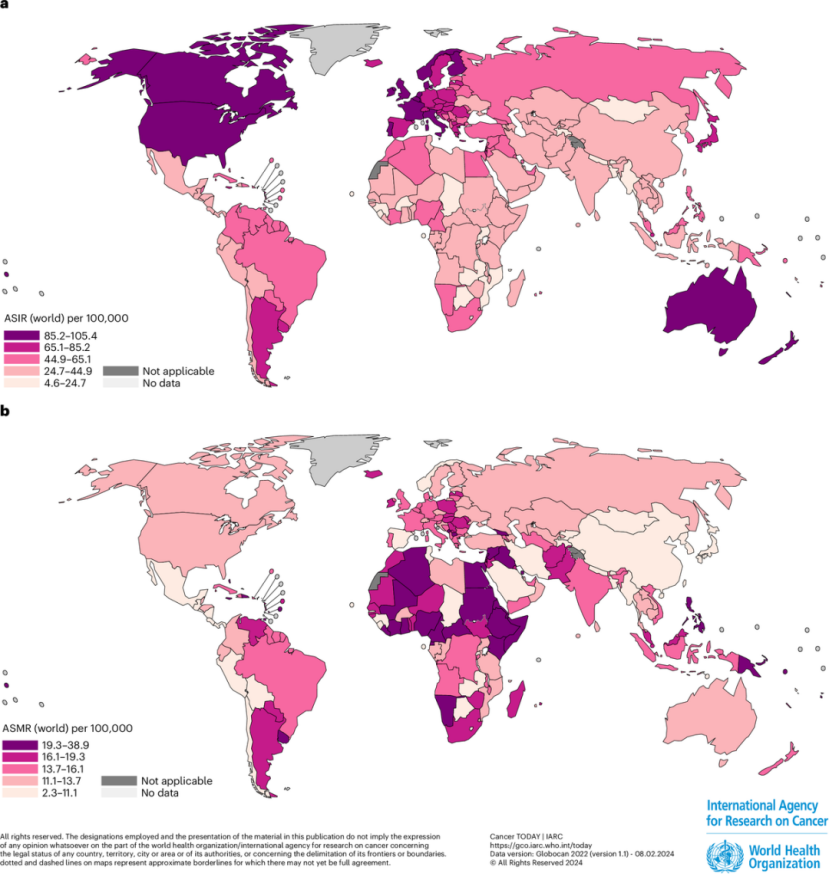
图1. 2022年全球女性按年龄段标准化乳腺癌发病率和死亡率地图[4]
1. 乳腺癌的分期与分型
乳腺癌的分期主要依据TNM分期法:
T(原发肿瘤)代表肿瘤大小及侵犯范围,从 Tis(原位癌)到 T4(肿瘤穿透胸壁或皮肤)逐步进展;
N(区域淋巴结)反映淋巴结受累程度,从 N0(无淋巴结转移)到 N3(锁骨上淋巴结或腋下深层淋巴结转移)逐级加重;
M(远处转移)提示是否发生远端转移,M0 为无转移,M1 为有转移。 综合三者可将乳腺癌分为 0 期(原位癌)、I/II 期(早期)、III 期(局部晚期)和 IV 期(晚期)。
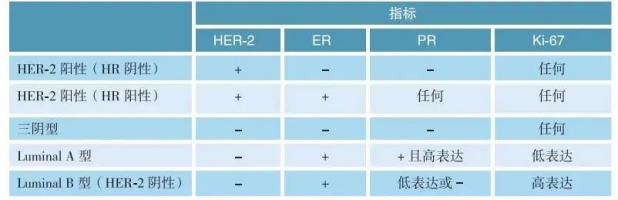
乳腺癌的分型基于雌激素受体(ER)、孕激素受体(PR)和人表皮生长因子受体-2(HER-2)的表达,具体分为:
Luminal A型:ER+/PR+,HER-2-,预后较好;
Luminal B型:ER+/PR+,伴 HER-2 + 或高增殖状态,预后介于 Luminal A 型与三阴性乳腺癌之间,治疗需结合 HER-2 状态综合制定;
三阴性乳腺癌(TNBC):ER-/PR-/HER-2-,预后较差,以化疗为主要治疗手段;
HER-2 阳性(HR 阴性)型:HER-2+,ER-/PR-,对 HER-2 靶向治疗敏感,未接受靶向治疗者预后不良;
HER-2 阳性(HR 阳性)型:HER-2+,ER+/PR+,需兼顾靶向治疗与内分泌治疗。
乳腺癌分型[5]
在乳腺癌研究中,利用动物模型模拟临床患者进行临床实验是常用的方法,其中,小鼠模型因实验周期短、成本低、繁殖快等优势,成为临床研究中最常用的实验动物模型,以下将详细介绍乳腺癌研究中常用的小鼠模型类型。
2. 乳腺癌自发肿瘤小鼠模型
自然易感品系小鼠模型
部分小鼠因遗传背景差异,在自然状态下对乳腺癌呈现不同易感性。这些品系无需人工基因操作,仅通过特定饲养条件维持,经过一定周期后可自发形成乳腺癌。
例如,A/JGpt品系的经产雌鼠易患乳腺癌,可能与特定基因自然变异或表达调控异常导致的乳腺组织细胞增殖分化异常有关。C3H/HeJGpt品系小鼠同样具有乳腺癌自然易感性,未交配及交配的雌鼠年长后均可能发生乳腺癌。这些自然易感品系为研究乳腺癌遗传易感因素提供了理想模型。
转基因小鼠模型
在乳腺癌研究中,相较于自然易感模型,转基因模型能更精准模拟特定致癌基因驱动的肿瘤发生发展过程。
以FVB-MMTV-PyMT模型为例,将致癌基因PyMT置于小鼠乳腺肿瘤病毒(MMTV)启动子调控下,使其在乳腺组织特异性表达。PyMT蛋白激活多种细胞内信号通路,促进增殖并抑制凋亡,最终导致乳腺肿瘤自发形成。该模型的肿瘤发生过程与人类乳腺癌高度相似,具有良好的客观性和重复性,是乳腺癌研究领域的经典模型。
部分验证数据如下:
FVB-MMTV-PyMT小鼠模型
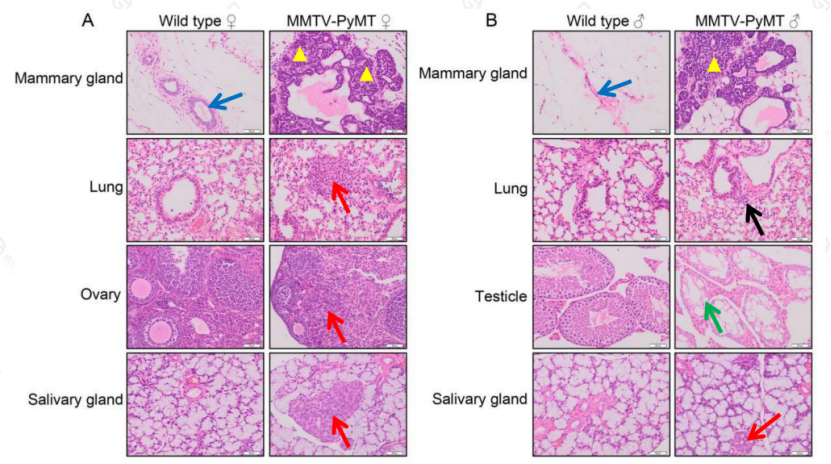
图2. MMTV-PyMT小鼠乳腺、肺及卵巢组织病理学检测
取MMTV-PyMT小鼠(6周龄)乳腺、肺、卵巢、唾液腺组织进行石蜡包埋、切片、HE染色,观察各组织病理变化(200x, bar=100 μm)。结果显示:雌鼠(图A):发展为乳腺癌,癌细胞组成实心团块或条索状癌巢;乳腺癌肺部转移,巢状病灶,炎性细胞浸润,卵巢中的肿瘤细胞多灶性生长,异型性明显;乳腺癌发生唾液腺转移,可见多个癌巢;雄鼠(图B):部分小鼠发展为乳腺癌,癌细胞组成实心团块或条索状癌巢;部分小鼠出现唾液腺转移;肺部支气管及肺泡间存在少量炎性病灶;睾丸中可见部分精曲小管坏死,精细胞脱落。Wild type小鼠乳腺、肺、卵巢未见明显异常。(注:黑色箭头:炎性细胞浸润,红色箭头:乳腺癌转移灶,蓝色箭头:乳腺导管,绿色箭头:曲精小管坏死,黄色三角区域:乳腺癌巢)
基因敲除小鼠模型
基因敲除模型聚焦于探究特定基因缺失对乳腺癌形成发展的影响,为解析发病机制提供关键研究视角。
亚星yaxing868官网通过敲除p53基因构建了B6-p53-KO模型。作为重要的抑癌基因,p53参与细胞周期调控、DNA损伤修复及凋亡过程。p53缺失会破坏基因组稳定性,提升细胞癌变概率,促使小鼠乳腺组织更易发生恶性转化并自发形成肿瘤。条件性基因敲除品系则是通过基因工程技术,实现目标基因在特定组织/细胞类型或时间点的敲除。该模型需与组织特异性工具鼠(如携带Cre重组酶的小鼠)交配以诱发肿瘤。
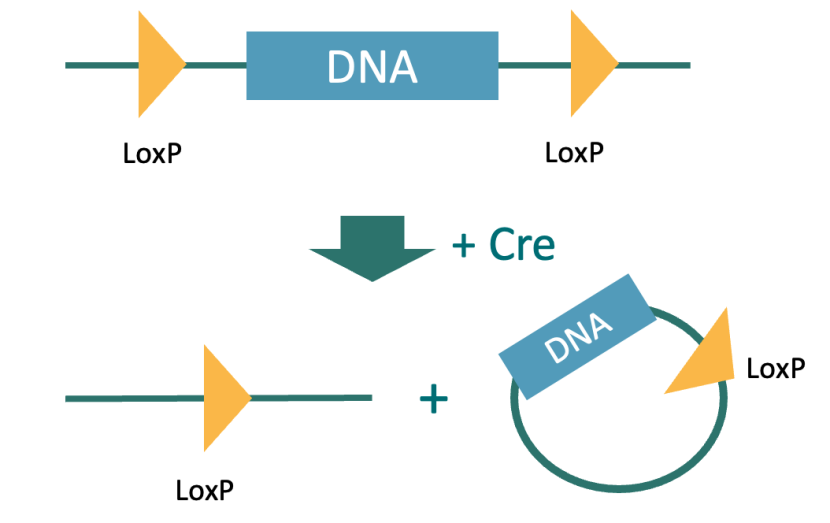
图3.条件性基因敲除
Brca1和Brca2作为重要的抑癌基因,在DNA损伤修复及基因组稳定维持中发挥关键作用。其条件性敲除会导致乳腺组织基因组不稳定,增加癌变风险。亚星yaxing868官网的Brca1-flox和Brca2-flox小鼠携带loxP位点,与H11-MMTV-iCre工具鼠交配后,iCre重组酶可在乳腺组织中特异性表达,进而介导Brca1或Brca2基因敲除。
亚星yaxing868官网部分乳腺癌自发肿瘤小鼠模型
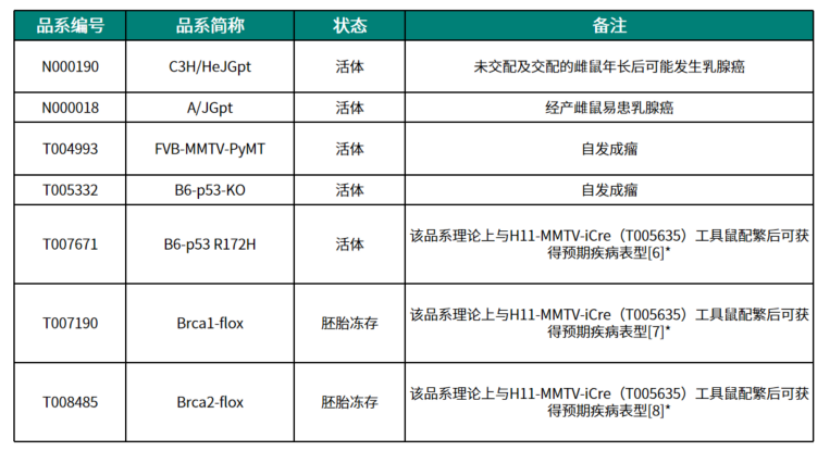
*部分品系尚未在乳腺组织中进行系统性验证实验,目前仅基于相关文献研究证实相关致癌/抑癌基因可诱发乳腺癌,建议在开展相关研究时谨慎评估模型适用性。
3. 乳腺癌移植瘤小鼠模型
除自发肿瘤模型外,移植瘤模型同样是乳腺癌研究的常用工具,主要包括异种移植瘤模型和同系移植瘤模型。相较于自发模型,移植瘤模型具有个体差异小、成瘤周期短、肿瘤细胞特性稳定可控等优势,在乳腺癌发生机制、药物筛选及疗效评估等方面具有重要作用。
异种移植瘤小鼠模型
乳腺癌异种移植瘤模型是将人类乳腺癌细胞系(CDX)或患者乳腺癌组织(PDX)移植到免疫缺陷小鼠体内形成肿瘤模型。在乳腺癌发生机制、药物筛选及疗效评估等研究中应用广泛。
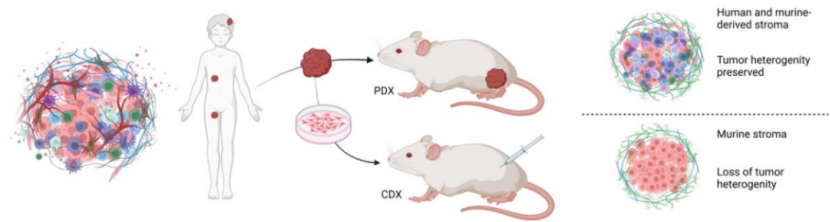
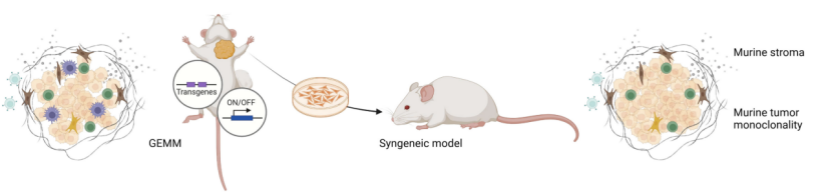
图4.异种移植瘤小鼠模型构建[9]
人源肿瘤组织异种移植小鼠模型(PDX 模型)
PDX模型将乳腺癌患者的肿瘤组织直接移植到免疫缺陷小鼠体内建立。该模型能较好保留原发肿瘤的组织学结构、遗传学特征和异质性,对肿瘤生物学行为、药物疗效预测等具有重要价值,但存在建立过程复杂、成本高、肿瘤可能适应宿主环境等问题。
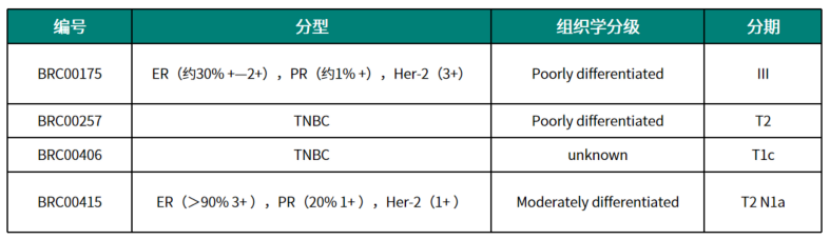
亚星yaxing868官网为满足疾病机理研究、药效测试、临床治疗对照的需求,已建立包含乳腺癌在内的多种常见癌症PDX资源库。
亚星yaxing868官网部分PDX资源
细胞系来源的异种移植瘤模型(CDX 模型)
CDX模型通过将人类乳腺癌细胞系接种免疫缺陷小鼠(如BALB/c裸鼠、NOD-Scid小鼠、NCG小鼠等)构建。该模型具有细胞系来源稳定、成本较低、实验周期短等显著优势,尤其适用于高通量药物筛选等研究;但其局限性在于无法完全再现人体肿瘤微环境,与人类肿瘤的复杂性存在差距。
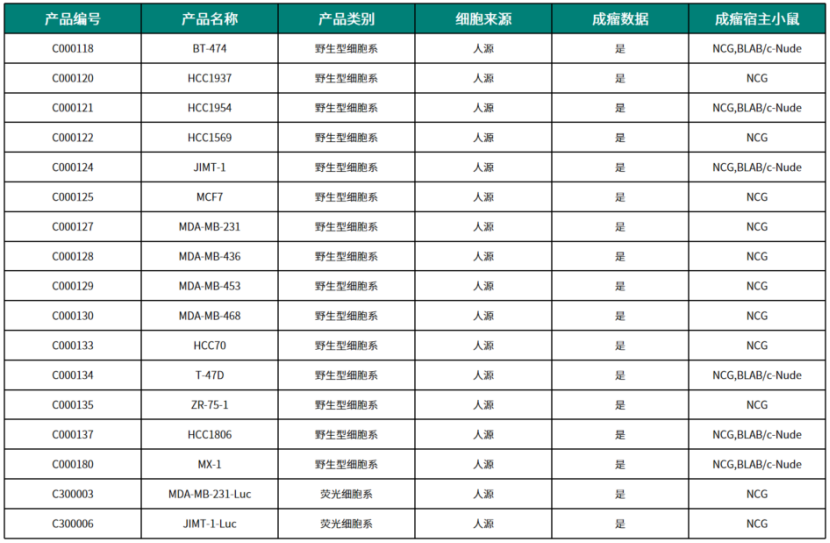
亚星yaxing868官网构建了完善的人源肿瘤细胞资源平台,涵盖野生型和荧光标记细胞系等,可充分满足各类行业研究的需求。
亚星yaxing868官网部分CDX资源
部分验证数据:
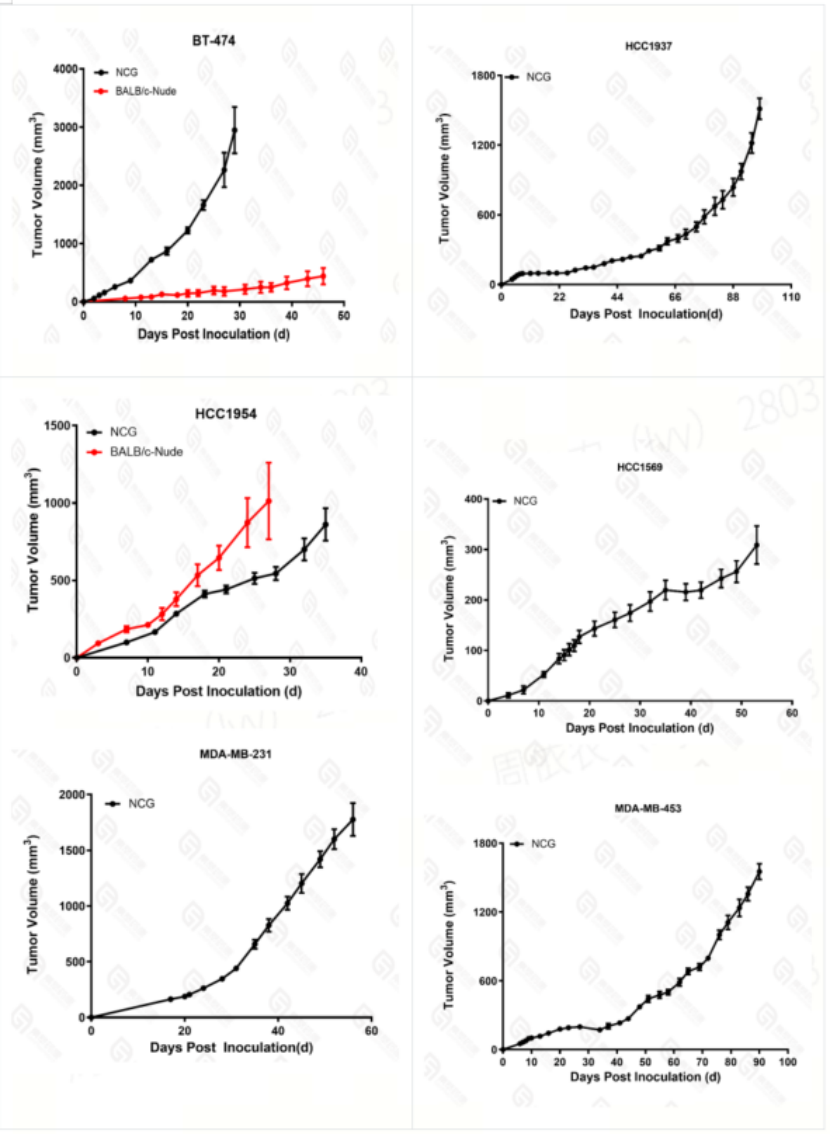
图5. 人源乳腺癌细胞系在免疫缺陷鼠中的成瘤情况
同系移植瘤小鼠模型
同系移植瘤模型是指将鼠源肿瘤细胞接种到具有相同遗传背景的免疫健全小鼠体内(如将4T1细胞接种到BALB/c小鼠)。该模型因保留完整的小鼠免疫系统,可用于研究肿瘤与免疫系统的相互作用,对免疫治疗研究具有重要意义;但由于肿瘤细胞异质性相对较低,与人类乳腺癌的复杂性仍存在差异。
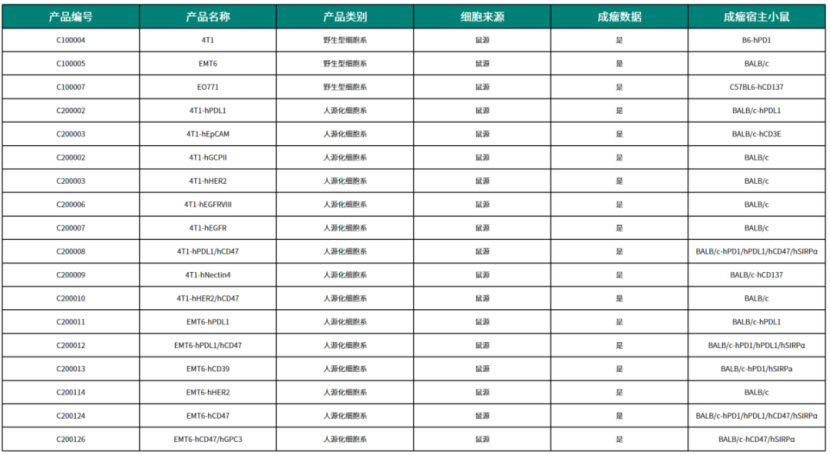
同系移植瘤移植物资源
亚星yaxing868官网构建了丰富的鼠源乳腺癌细胞资源库,涵盖野生型、人源化、荧光标记细胞系等,为企业实验研究提供支撑,助力药物研发进程。
亚星yaxing868官网部分同系移植瘤移植物资源
部分验证数据:
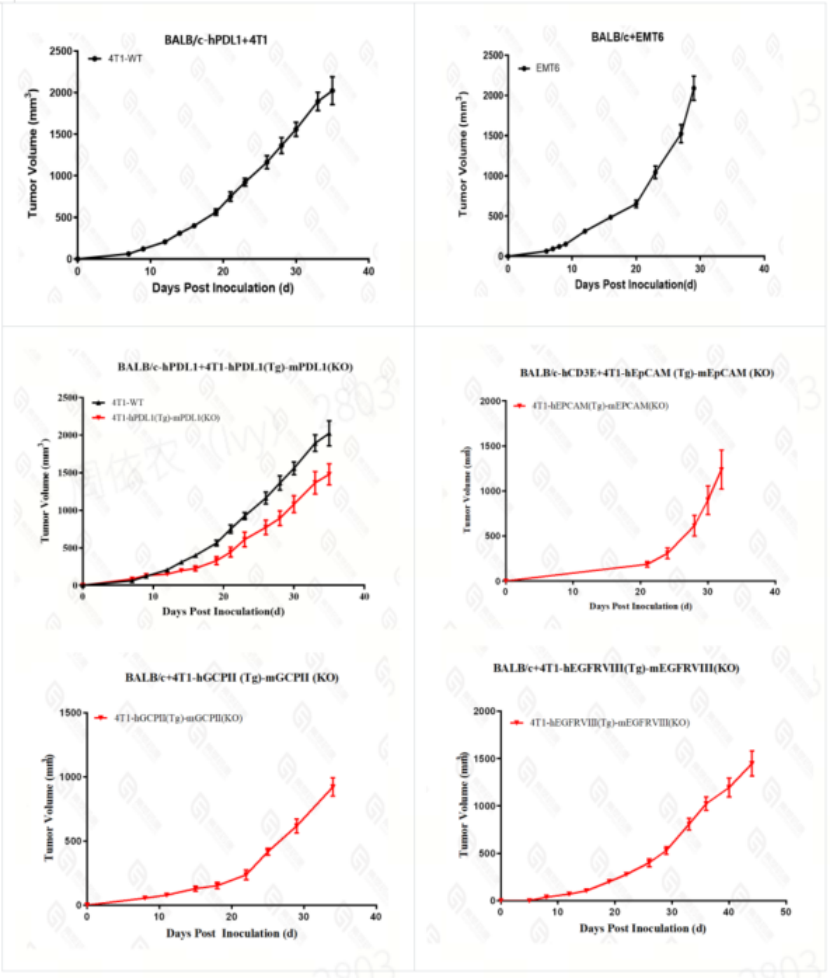
图7.鼠源乳腺癌细胞系在免疫健全鼠中的成瘤情况
同系移植瘤宿主资源:乳腺癌药物靶点人源化小鼠模型
在乳腺癌药物研发领域,Her2、Egfr、Vegfa等热门靶点持续引领创新方向。靶点人源化小鼠模型通过将小鼠靶点替换为人类同源基因,有效弥补了啮齿类动物模型与临床之间的差异,成为科研探索的重要工具。科研人员可以利用该类模型开展药物安全测试,观察药物对人源化小鼠的正常器官组织产生靶向作用,这有助于筛选出潜在安全性风险较高的药物,降低后续研发的成本和时间损耗。而药物靶点人源化小鼠模型与乳腺癌细胞系的协同应用,更是为乳腺癌药物研发构建了从体外筛选到体内验证的完整技术链条,不仅加速了新型靶向药物的研发进程,提升了药物研发成功率,更有望为乳腺癌患者带来更多有效的治疗选择。
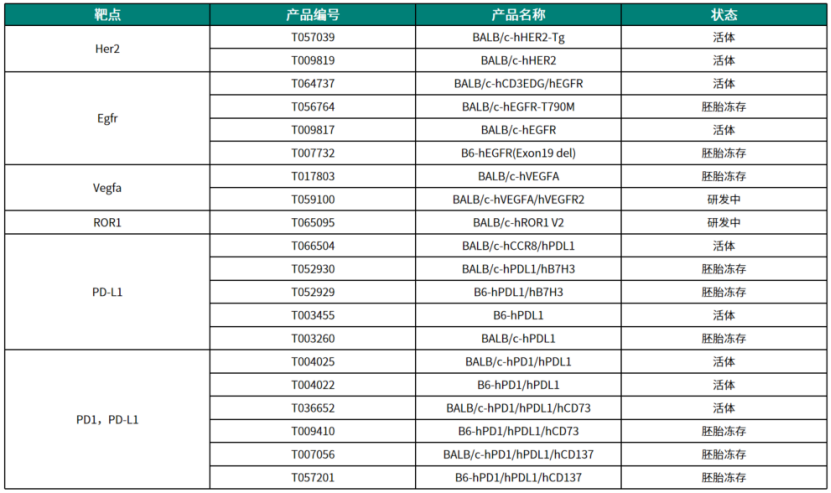
亚星yaxing868官网部分乳腺癌药物靶点人源化模型
*文中涉及的组织/细胞资源仅限于亚星yaxing868官网相关服务项目,不支持单独出售;如需获取具体小鼠模型数据,欢迎发送邮件至marketing@gempharmatech.com咨询。
参考文献
[1]CA Cancer J Clin. 2024 May-Jun;74(3):229-263.
[2]Ann Transl Med. 2022 Aug;10(15):813.
[3]J Oncol. 2021 Nov 30;2021:4891936.
[4]Nat Med. 2025 Apr;31(4):1154-1162.
[5]CSCO乳腺癌诊疗指南. 2024
[6]Journal of Pharmacology and Experimental Therapeutics. 2023 May 18;2023:43-43.
[7]Br J Cancer. 2009 Sep 29;101:1651–1657.
[8]Oncogene. 2001 Aug;20(30):3937-48.
[9]Front Oncol. 2024 Feb 2;14:1333129.

